Mécanismes de la traduction

Traduction ribosomale archée et évolution
L'étude des processus fondamentaux, comme la biosynthèse des protéines, permet de mieux comprendre l'évolution. Ainsi, l'analyse des séquences de l’ARN ribosomal de la petite sous-unité a conduit à définir un arbre du vivant organisé en trois domaines, bactéries, eucaryotes et archées. Au sein de cet arbre les archées sont proches des eucaryotes et apparaissent comme une pièce centrale pour comprendre l'évolution des organismes vivants. Les efforts pour mieux comprendre la diversité des archées et de leurs mécanismes moléculaires apportent des informations importantes.
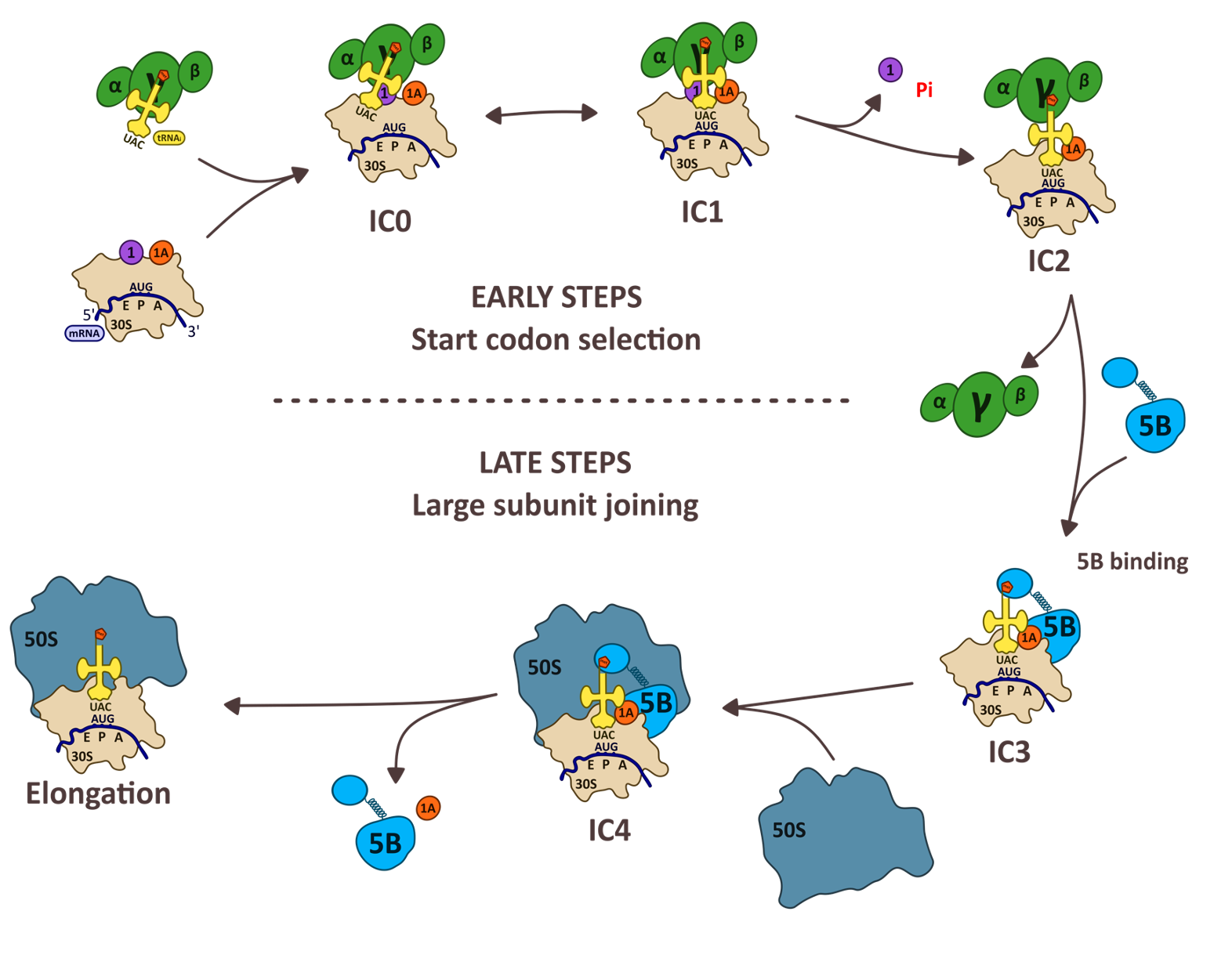
Chez les archées, les ARNm ne sont pas modifiés après leur transcription, comme chez les bactéries. Selon le phylum d’archée, les ARNm portent des séquences Shine-Dalgarno ou ont des régions 5' non traduites très courtes (sans leader). Cependant, d'autres caractéristiques de l'appareil de traduction rapprochent les archées des eucaryotes. En particulier, le ribosome archéen et les facteurs de traduction sont de type eucaryote. Notre équipe s'intéresse principalement aux complexes ribosomaux d'initiation de la traduction. Nous étudions les organismes archéens et nous nous intéressons également aux bactéries et aux eucaryotes pour mieux comprendre comment les mécanismes d'initiation de la traduction ont évolué. Nous utilisons la cristallographie aux rayons X, le SAXS et la cryo-EM pour réaliser des études structurales et nous avons également développé des techniques biochimiques pour les études fonctionnelles.
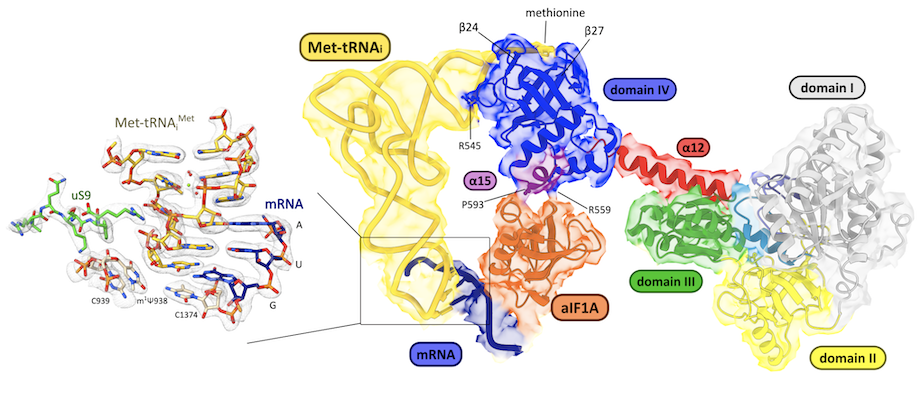
Le ribosome des archées est de type eucaryote. Les archées appartenant aux branches des TACK et des Asgards, les plus proches parents des eucaryotes, ont des ribosomes contenant des protéines ribosomales eucaryotes que l'on ne trouve pas dans les autres archées, eS25, eS26 et eS30. Récemment, nous avons étudié le cas de Saccharolobus solfataricus, une TACK archaea, qui a en plus la particularité de posséder majoritairement (72%) des ARNm sans leader. Nous avons déterminé des structures par cryo-EM de la petite sous-unité ribosomique de S. solfataricus liée soit à des ARNm possédant des séquences SD ou à des ARN messager sans leader. Les structures cryo-EM montrent que eS25, eS26 et eS30 sont liées à la petite sous-unité comme elles le sont chez les eucaryotes.
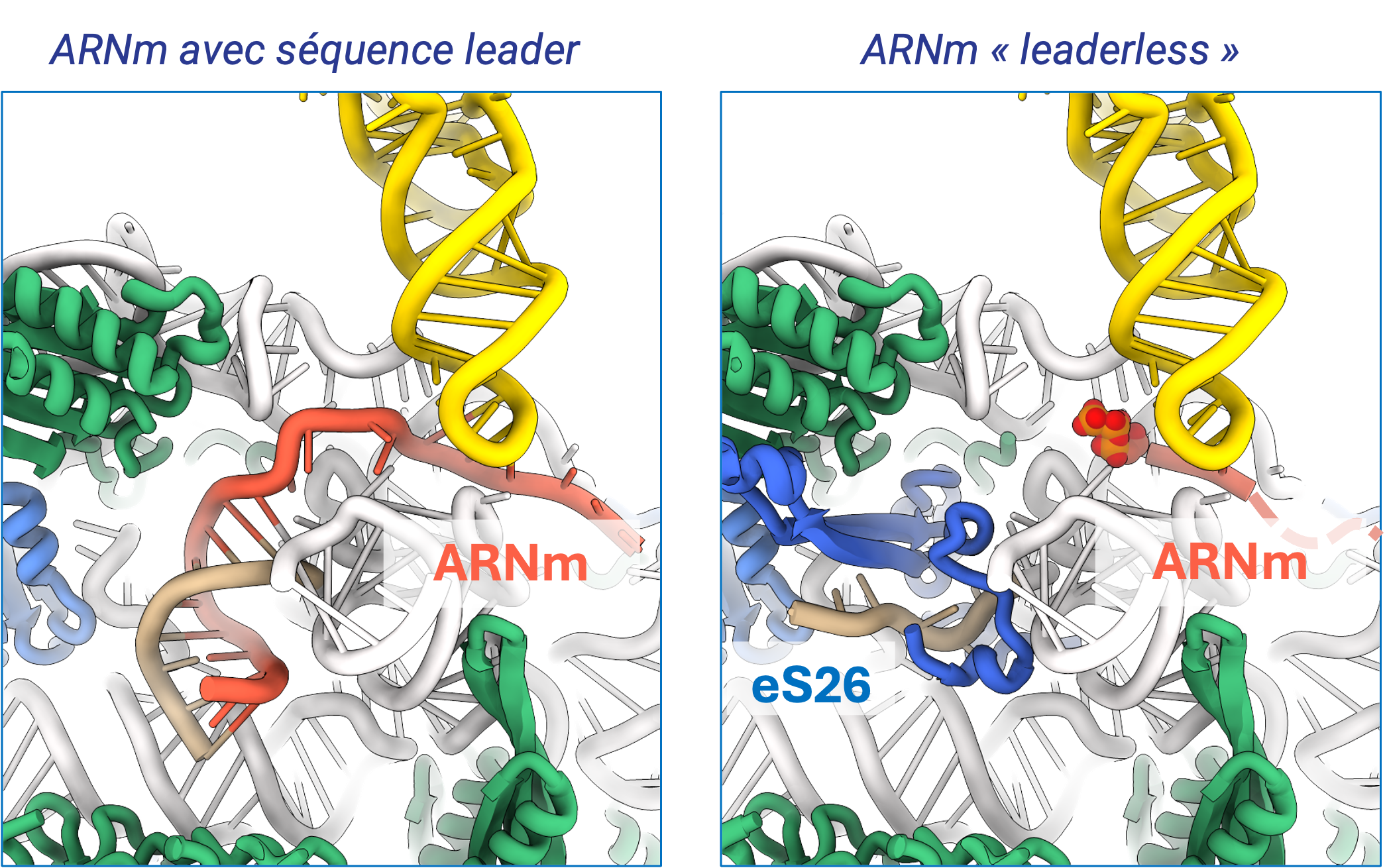
Nous identifions deux protéines ribosomiques, aS33 et aS34, et un domaine supplémentaire de la protéine eS6. Les ARNm sans leader sont liés à la petite sous-unité avec la contribution de leur groupement 5'-triphosphate. Le domaine eS26 de l'archée se lie au canal de sortie de l'ARNm enroulé autour de l'extrémité 3' de l'ARNr, comme cela est observé chez les eucaryotes. Sa position n'est pas compatible avec celle du duplex SD:antiSD. Nos résultats suggèrent un rôle positif d’eS26 dans la traduction des ARNm sans leader et des voies évolutives possibles de la traduction archéenne à la traduction eucaryote (Bourgeois G, et. al, Nat commun 2025).
D'autres modèles cellulaires archées, bactériens et eucaryotes sont également étudiés dans l'équipe pour mettre à jour les spécificités des mécanismes de traduction.
Ingénierie des protéines
L'incorporation spécifique d'un acide aminé beta dans un polypeptide introduirait une flexibilité sans précédent dans la chaîne principale, élargissant ainsi les possibilités géométriques de repliement des protéines. Il a récemment été démontré, à l'aide d'un système de traduction in vitro sans cellules, que le ribosome était capable d'incorporer efficacement un acide aminé beta dans un polypeptide. Ainsi, pour permettre la synthèse in vivo de protéines contenant des acides-aminés beta il reste à concevoir des aminoacyl-ARNt synthétases capables d'estérifier efficacement ces acides aminés beta sur un ARNt spécifique. Nous avons travaillé sur ce problème en collaboration avec Philippe Marlière (Heurisko INC, USA) et Valérie Pezo (ISSB, Evry) en utilisant la méthionyl-tRNA synthétase (MetRS) de E. coli. Les études biochimiques et structurales de la liaison de la méthionine à la MetRS donnent des indices intéressants pour renforcer l’activité catalytique de l’enzyme vis-à-vis des acides aminés beta, avec notamment une structure cristallographique à 1,4 Å de résolution pour un complexe MetRS:beta-Met. D'autres projets d'ingénierie de protéines sont en cours.
eIF2 dans les maladies cardiaques
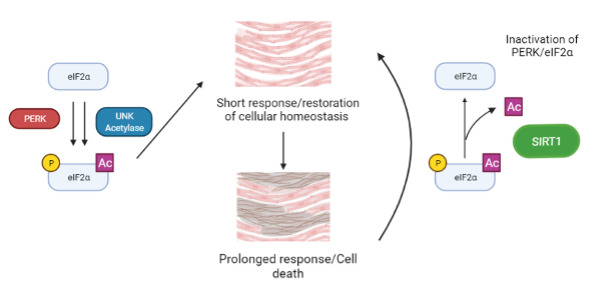
Le stress du réticulum endoplasmique (RE) est apparu comme un mécanisme important impliqué dans la pathogenèse des maladies cardiovasculaires. eIF2 est un facteur impliqué dans le démarrage de la traduction, mais il est aussi un effecteur clé de la réponse au stress du RE. La perturbation de la fonction du RE entraîne une accumulation de protéines, ce qui déclenche la « réponse aux protéines non repliées » (UPR). Au cours de cette réponse, eIF2 est phosphorylé sur sa sous-unité alpha par la kinase PERK. Cela déclenche une série d'événements de transduction pour réduire l'accumulation de protéines et le stress du RE. Une induction légère à modérée de cette voie favorise une réponse restauratrice alors qu'une réponse sévère ou chronique au stress du RE favorise l'apoptose et contribue au développement de la maladie cardiaque.
Une découverte récente de notre collaborateur, C. Lemaire (INSERM UMR-S1180), a montré que cette voie était contrôlée non seulement par la phosphorylation d'eIF2, mais aussi par son acétylation. De plus, la désacétylation d'eIF2 par la protéine sirtuin 1 (SIRT1), protège les cardiomyocytes de l'apoptose induite par le stress ER. La voie SIRT1-eIF2 pourrait être utilisée en thérapie médicamenteuse, car les taux d'activité de SIRT1 peuvent être modifiés par des composés activateurs de sirtuines (STACs). Notre objectif est de comprendre les bases moléculaires de l'interaction de SIRT1 avec eIF2α, ainsi que l'interaction entre l'acétylation et la phosphorylation d'eIF2α.
Collaborations :
Laurence Blanchard et Arjan de Groot, BIAM CEA Cadarache
Franck Martin, UPR 9004, IBMC Strasbourg
Béatrice Clouet D’Orval, CBI, Toulouse
Didier Flament, IFREMER Brest
SOLEIL Synchrotron
Anh Tuân Phan (NTU, Singapore)
Philippe Marlière (Exapto, Palaiseau), Valérie Pezo (ISSB, Evry)
Christophe Lemaire, INSERM UMR-S1180
Sélection de publications :
Paupelin-Vaucelle H, Boschiero C, Lazennec-Schurdevin C, Schmitt E, Mechulam Y, Marlière P, Pezo V. Cys-tRNAj as a Second Translation Initiator for Priming Proteins with Cysteine in Bacteria. ACS Omega. 2025 J10(5):4548-4560. doi.org/10.1021/acsomega.4c08326. eCollection 2025
Bourgeois G, Coureux PD, Lazennec-Schurdevin C, Madru C, Gaillard T, Duchateau M, Chamot-Rooke J, Bourcier S, Mechulam Y, Schmitt E. Structures of Saccharolobus solfataricus initiation complexes with leaderless mRNAs highlight archaeal features and eukaryotic proximity. Nat Commun 2025 16: 348 doi.org/10.1038/s41467-024-55718-5
Fedry J, Silva J, Vanevic M, Fronik S, Mechulam Y, Schmitt E, des Georges A, Faller WJ, Förster F. Visualization of translation reorganization upon persistent ribosome collision stress in mammalian cells. Mol Cell. 2024 Mar 21;84(6):1078-1089.e4, doi.org/10.1016/j.molcel.2024.01.01
Opuu V, Nigro G, Lazennec-Schurdevin C, Mechulam Y, Schmitt E, Simonson T. Redesigning methionyl-tRNA synthetase for β-methionine activity with adaptive landscape flattening and experiments.Protein Sci. 2023 Sep;32(9):e4738, doi.org/10.1002/pro.4738
Cardenal Peralta C, Vandroux P, Neumann-Arnold L, Panvert M, Fagart J, Seufert W, Mechulam Y, Schmitt E. Binding of human Cdc123 to eIF2γ. J Struct Biol. 2023 Sep;215(3):108006, doi.org/10.1016/j.jsb.2023.108006.
Kazan R, Bourgeois G, Lazennec-Schurdevin C, Larquet E, Mechulam Y, Coureux PD, Schmitt E. Role of aIF5B in archaeal translation initiation. Nucleic Acids Res. 2022, 50, 6532–6548, doi.org/10.1093/nar/gkac490.
Nigro G, Bourcier S, Lazennec-Schurdevin C, Schmitt E, Marlière P, Mechulam Y. Use of β3-methionine as an amino acid substrate of Escherichia coli methionyl-tRNA synthetase. J Struct Biol. 2020, 209, 107435, doi.org/10.1016/j.jsb.2019.107435.
Coureux PD, Lazennec-Schurdevin C, Bourcier S, Mechulam Y, Schmitt E. Cryo-EM study of an archaeal 30S initiation complex gives insights into evolution of translation initiation. Commun Biol. 2020, 3, 58, doi.org/10.1038/s42003-020-0780-0.
Schmitt, E, Coureux, P-D, Kazan, R., Bourgeois, G., Lazennec-Schurdevin, C., Mechulam, Y. Recent advances in archaeal translation initiation. Frontiers in Microbiology, section Biology of Archaea. Front Microbiol. 2020 Sep 18;11:584152, doi.org/10.3389/fmicb.2020.584152.




