Fonctions des modifications des ARN et des protéines
Fonctions des modifications des ARN et des protéines
La plupart des ARN et des protéines participant à la maturation et à la traduction des ARNm sont modifiés de manière post-transcriptionnelle ou post-traductionnelle par de nombreuses modifications chimiques petites et diverses, qui améliorent l'efficacité, la fidélité et la régulation de processus biologiques fondamentaux tels que la maturation, la stabilité ou la traduction des ARNm.
Ces modifications sont largement conservées au cours de l'évolution, mais leur rôle a longtemps été négligé en raison de l'absence fréquente de phénotypes évidents dans les organismes unicellulaires ou de la nature encore inconnue des enzymes (ou writers) qui ajoutent ces marques. Au cours de la dernière décennie, de nombreuses études ont démontré le rôle critique de ces modifications. Les writers apparaissent aujourd'hui comme essentiels pour plusieurs processus cellulaires ainsi que pour des pathologies humaines telles que les cancers et les troubles du développement neurologique.
L'équipe cherche à décrypter le rôle de ces modifications post-transcriptionnelles et post-traductionnelles dans des processus biologiques centraux (maturation, traduction et décroissance des ARNm), dans la prolifération cellulaire, dans le développement des organes et à décrire les conséquences de mutants pathogènes sur les fonctions des enzymes responsables de ces modifications.
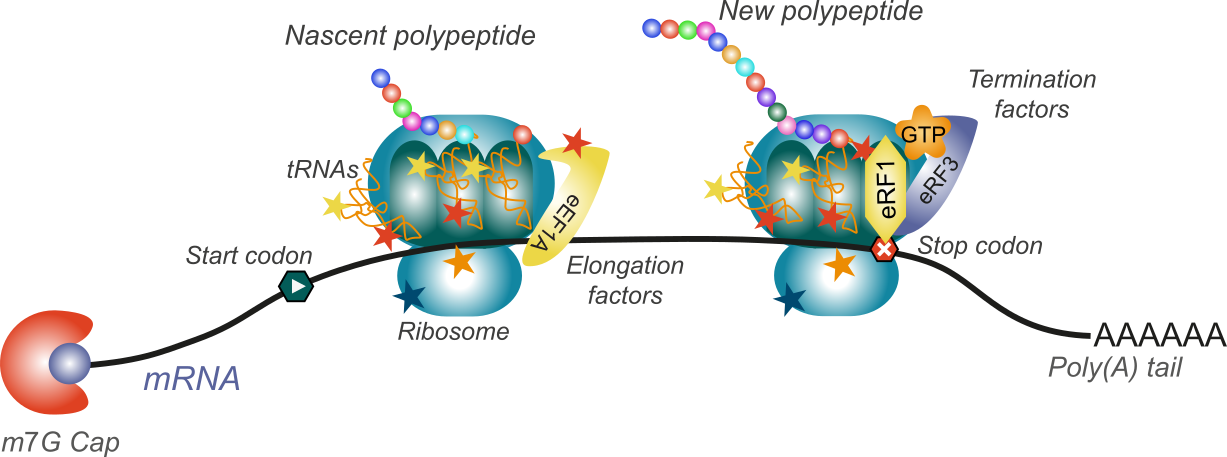
Les modifications des ARN et des protéines au coeur de la synthèse des protéines
(Artwork : Morgane Corre)
L'équipe utilise de nombreuses approches expérimentales, ce qui est rendu possible grâce aux infrastructures locales mises en place au BIOC, pour la biologie moléculaire, l'expression et la purification des protéines, la biologie structurale, la biologie cellulaire, la biochimie des ARN...

Quelques techniques utilisées au laboratoire
La protéine TRMT112, un activateur majeur de méthyltransférases
L’équipe étudie depuis de très nombreuses années, la protéine TRMT112 qui est présente dans les trois domaines du vivant et qui agit comme un régulateur allostérique de plusieurs méthyltransférases (MTases). Nous nous intéressons tout particulièrement à la protéine TRMT112 des organismes eucaryotes, qui interagit avec des MTases modifiant des facteurs (ARNt, ARNr, protéines…) impliqués dans les processus de maturation et de traduction des ARN messagers (ARNm) (Wang et al; 2024).
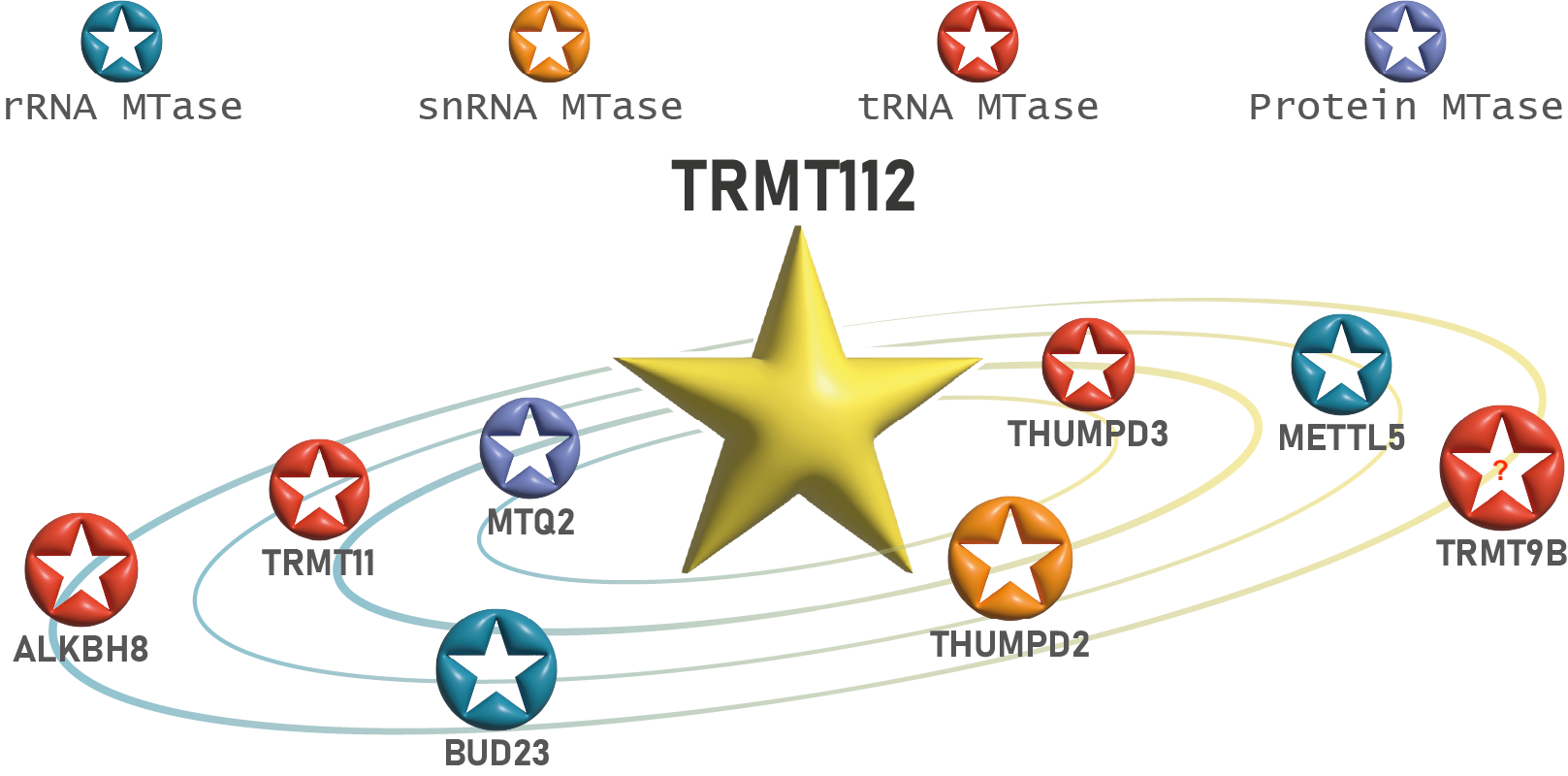
Le réseau d'interaction de la protéine TRMT112 humaine.
(Artwork : Morgane Corre)
Chez l’humain, les complexes TRMT112-BUD23 et TRMT112-METTL5 catalysent la formation de N7-méthylguanosine (m7G) et de N6-méthyladénosine (m6A) sur l'ARNr 18S, respectivement et participent à la voie de biogenèse de la sous-unité 40S du ribosome. Le complexe TRMT112-MTQ2 cible le facteur de terminaison de la traduction eRF1, qui est essentiel pour la libération des protéines nouvellement synthétisées. Les complexes TRMT112-ALKBH8, TRMT112-THUMPD3 et TRMT112-TRMT11 contribuent à l'élongation de la traduction en modifiant les ARNt. Le complexe TRMT112-THUMPD2 modifie le petit ARN nucléaire U6 (snRNA U6) qui est impliqué dans l’épissage des ARN pré-messagers. Enfin, les fonctions moléculaires et biologiques précises du complexe TRMT112-TRMT9B restent à élucider. Certains de ces complexes sont également présents chez certaines archées, ce qui souligne les fortes similarités entre les machineries de synthèse protéique chez les eucaryotes et les archées.
Grâce à un large panel d’approches et avec l’aide de différents collaborateurs, l’équipe a étudié le réseau d’interaction des protéines TRMT112 humaine, de la levure Saccharomyces cerevisiae ou d’archées avec différentes MTases. Nous avons découvert les fonctions biochimiques des complexes TRMT112-METTL5, TRMT112-THUMPD3 et TRMT112-THUMPD2. Nous avons également caractérisé les activités enzymatiques de tous ces complexes et avons obtenu des informations structurales sur plusieurs de ces complexes. Ceci a révélé que ces différentes MTases interagissent par un mode très similaire avec TRMT112 malgré une faible identité de séquence (moins de 20%) et sont donc directement en compétition les unes avec les autres. Ces études ont permis de préciser les différents rôles joués par TRMT112 pour activer ces MTases. En effet, TRMT112 stabilise ces MTases in vivo et in vitro, leur confère une capacité de fixation de la S-adenosyl-L-méthionine (SAM), le groupement donneur de méthyle utilisé par toutes ces MTases et contribue aussi directement à la fixation du substrat par certains complexes.
Comme la dérégulation des niveaux d’expression de la protéine TRMT112 ou des MTases associées affectent la prolifération de cellules cancéreuses et comme des mutations pathogènes associées à des troubles du développement neurologique ont été identifiées dans plusieurs MTases interagissant avec TRMT112, l’équipe a pour objectif de mettre à jour les fonctions biologiques de ces MTases et leur importance dans le développement du système nerveux.



